EL CÓDIGO DE BARRAS DE LA VIDA COMO UNA HERRAMIENTA PARA LA IDENTIFICACIÓN DE LARVAS DE PECES
Saludos cordiales amigos de Steemit, comunidad de @SteemStem, @Stem-espanol, @utopian-io, @curie. Para complementar la publicación anterior sobre estadios tempranos de peces, aquí les traigo cómo utilizar el código de barras del ADN para identificar las larvas. Para ello, voy a destacar a manera de ejemplo el caso de la familia Labrisomidae y el uso de secuencias del gen que codifica para la enzima Citocromo Oxidasa I (COI) como marcador.
INTRODUCCIÓN
El empleo de marcadores moleculares para la determinación de larvas de peces, implica la necesidad de un adulto claramente identificado con el cual comparar. De esta manera, pierden efectividad para evaluar un lote de larvas capturado directamente en el campo si no hay un adulto con una apropiada determinación taxonómica sobre el cual se haya empleado el mismo marcador, por lo que en algunas de ellas sólo se podrá llegar morfológicamente hasta familia. Esta problemática no se presenta sólo con larvas, sino también para la identificación de material biológico en general. Por ejemplo, para la determinación de la identidad de fragmentos de organismos, certificación de origen de productos alimenticios ya procesados o crudos, restos óseos y cualquier material biológico que amerite conocerse su determinación específica. En este sentido, la multiplicidad de opciones moleculares desarrolladas en los últimos años (Avise, 2004), complica la posibilidad de tener una base de datos amplia que sirva como herramienta para la identificación de especies.
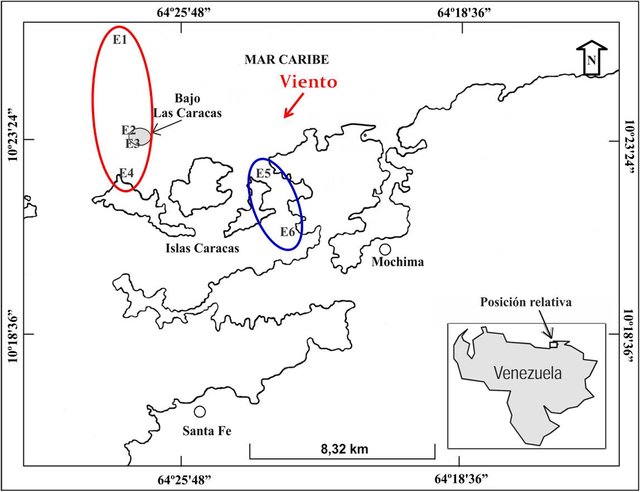
Figura 1. Localización de las estaciones de muestreo en el estado Sucre, Venezuela.
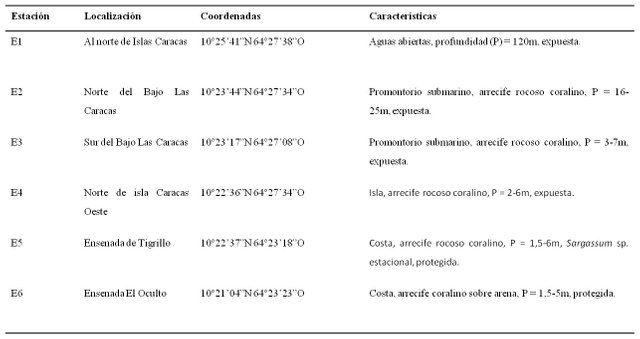
Tabla 1. Estaciones de muestreo, localización geográfica y principales características de las estaciones.
Figura 2. Fotografías en las que se destaca la estructura de los fondos de arrecifes sobre los cuales se realizaron muestreos de ictioplancton. A: Arrecifes fundamentalmente rocosos con corales pétreos y octocorales, B: Arrecife fundamentalmente rocoso con corales pétreos, pero con desarrollo estacional de Sargassum sp., C: Arrecife fundamentalmente coralino, sobre un sustrato arenoso.
Para la recolección de muestras de ictioplancton se hicieron arrastres, entre 0,5 y 3m de profundidad, utilizando una red estándar para captura de ictioplancton (Holden y Raitt, 1975; Boltowskoy, 1999), con un diámetro de boca de 62cm y 500µm de abertura de malla. Los arrastres se efectuaron a una velocidad promedio de 2 nudos, durante 15 minutos en cada una de las estaciones. La red estuvo equipada con un medidor de flujo para estandarizar los datos de abundancia a densidad (Fig. 3).
Figura 3. Fotografías del método de muestreo utilizado y del tamiz colector del plancton.
El ADN de las larvas se aisló a partir de cada espécimen completo obtenido de los arrastres durante el muestreo de plancton, mediante el empleo del Kit “NucPrep™ Chemistry Isolation” (Applied Biosystems, Inc.), siguiendo un procedimiento previo de digestión, para luego pasar a las fases de prefiltración, purificación y elución, empleando el equipo “ABI PRISM™ 6100 Nucleic Acid PrepStation” (Applied Biosystems, Inc.).
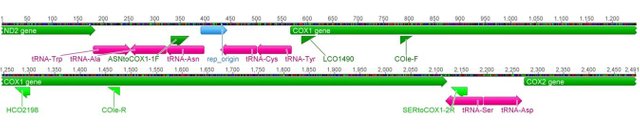
Debido a la heterogeneidad taxonómica del material biológico analizado, para la amplificación por PCR (Polimerasa Chain Reaction) se utilizaron los juegos de cebadores LCO/HCO (5ˈ-GGT CAA CAA ATC ATA AAG ATA TTG G-3ˈ/ 5ˈ-TAA ACT TCA GGG TGA CCA AAA ATC A-3ˈ) (Folmer et al., 1994), COIeF/COIeR (ATA ATG ATA GGA GGR TTT GG / GCT CGT GTR TCT ACR TCC AT) (Arndt et al., 1996) y diversas combinaciones: COIeF/HCO, LCO/COIeR, COX1F/HCO, COX1F/COIeR (Fig. 4). Estos cebadores son necesarios para que la enzima ADN polimerasa pueda iniciar las copias del ADN.
Figura 4. Posición relativa de los diferentes cebadores empleados en la amplificación de los fragmentos del gen COI.
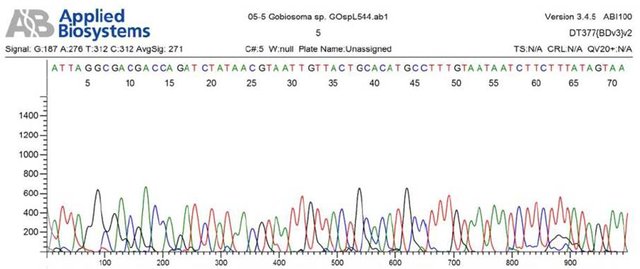
Los productos de las reacciones de PCR fueron enzimáticamente purificados para remover restos de cebadores y de dNTPs no consumidos durante la PCR. Luego, la reacción de secuenciación se llevó a cabo empleando el kit de secuenciación BigDye Terminator v.3.1 (Applied Biosystems, Inc.). Los productos de secuenciación fueron separados y detectados en un equipo ABI PRISM 377-XL. Los electroferogramas obtenidos fueron revisados mediante el programa BioEdit (Hall, 2011), a fin de corregir posibles errores en las secuencias (Fig. 5).
Figura 5. Revisión de los electroferogramas para corregir posibles errores en las secuencias
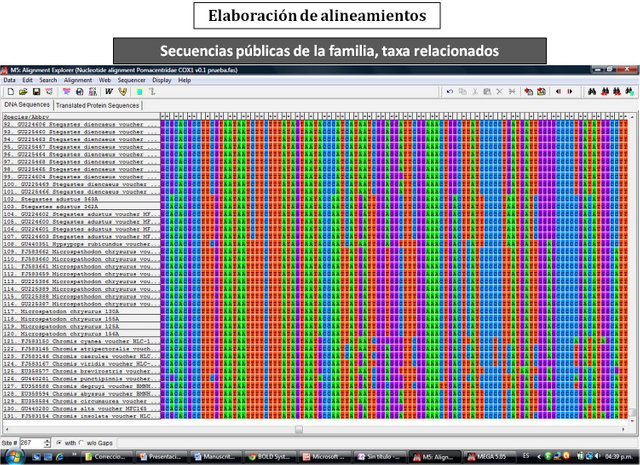
En un primer paso, la posición taxonómica tentativa de las muestras fue inferida comparando la secuencia problema con las bases de datos GenBank y de la iniciativa BOLD, a través de las aplicaciones respectivas: BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) y BOLD-IDS (http://www.boldsystems.org/views/idrequest.php). Esta ubicación taxonómica tentativa obtenida para cada muestra, permite su alineamiento con datos homólogos de taxones co-familiares, utilizando para ello tanto secuencias propias como aquellas disponibles en las bases de datos indicadas (Fig. 6).
Figura 6. Captura de pantalla de uno de los alineamientos para los análisis filogenéticos.
Los alineamientos fueron hechos con el programa MEGA versión 5 (Tamura et al., 2011), el cual fue empleado igualmente para las inferencias filogenéticas. Considerando que el objetivo de este trabajo no es inferir relaciones filogenéticas robustas sino ubicar sistemáticamente ejemplares con base en secuencias del gen COI, se asumió para todas las familias el modelo Kimura 2-Parametrós (Kimura, 1980) para la construcción de los árboles. Ello permite una uniformidad metodológica respecto a las estimaciones de BOLD, que usan este mismo modelo. La obtención de las topologías, mostrando las relaciones filogenéticas y la inclusión de las muestras problema monofiléticamente en un clado, fue llevada a cabo mediante el método de Neighbor-Joining (Saitou y Nei, 1987). Se realizó un test bootstrap de 2000 réplicas y cercano a cada rama del árbol se muestra el porcentaje de árboles (réplicas) en los cuales los taxones asociados se agrupan (Felsenstein, 1985).
A pesar de que buena parte de los lectores seguramente no se encuentran familiarizados con esta terminología, se colocó la información técnica metodológica, a fin de que cualquier especialista pueda hacer uso de ella.
RESULTADOS
Las secuencias del gen COI permitieron la identificación molecular de 45 especies de larvas de peces. Un número de 14 ejemplares de larvas sólo se pudieron ubicar en el nivel taxonómico de género, basándose en las secuencias COI obtenidas y las disponibles para comparación. La determinación a nivel de familia fue obtenida para 1 larva. Un número de 35 especies de larvas (75%), constituyeron nuevos aportes al conocimiento del desarrollo larvario de alguna especie, haciendo aportes fundamentales en tallas que van desde 1,5mm a 6,8mm, las cuales incluyen a larvas de muy pequeño desarrollo que hasta ahora no eran conocidas.
Por razones de espacio, en esta publicación muestro el aporte del código de barras en la identificación larvaria de ejemplares de la familia Labrisomidae. Los labrisómidos son un grupo de peces dentro del orden Perciformes, abundantes en aguas tropicales de los oceános Pacífico y Atlántico, con aletas dorsales grandes las cuales poseen mayor cantidad de espinas que de radios blandos (Fig. 7) (Nelson, 1994).
Figura 7. Fotografía de un ejemplar de Labrisomus nuchipinnis, en un arrecife del Golfo de Cariaco, estado Sucre, Venezuela.
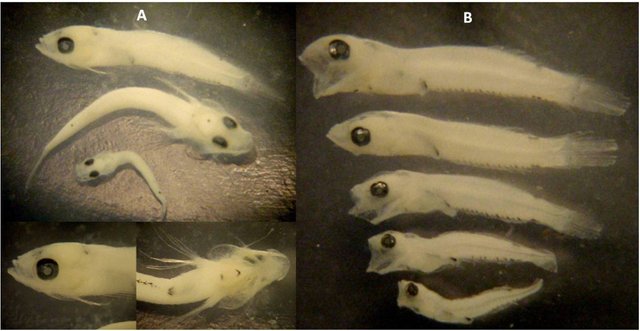
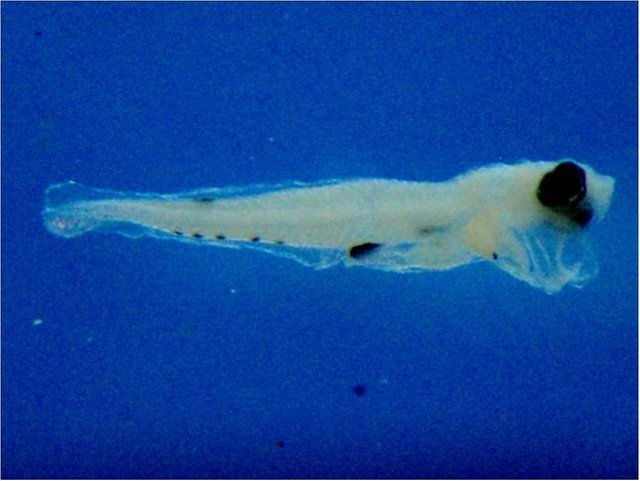
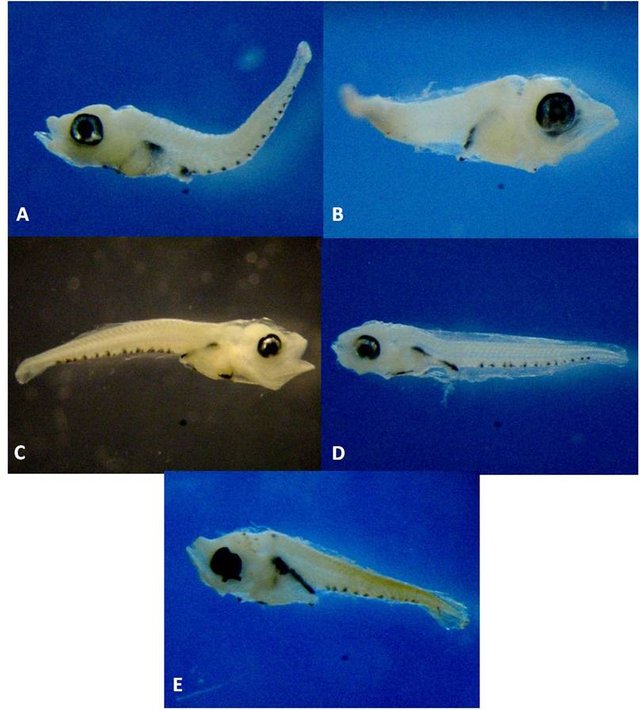
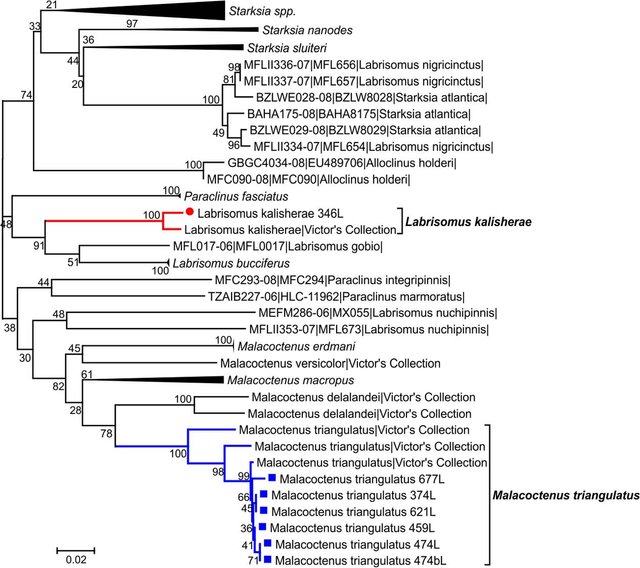
Para esta familia, fueron obtenidas secuencias COI a partir de 8 larvas, las cuales permitieron su asignación específica: 176L de Labrisomus kalisherae (Fig. 8); 374L (Fig. 9A), 459L (Fig. 9B), 474L (Fig. 9C), 621L (Fig. 9D), 762L (Fig. 9E) de Malacoctenus triangulatus. Todas estas secuencias fueron logradas a partir de amplificaciones empleando los primers LCO:HCO. El juego de datos para la elaboración del árbol contuvo 66 secuencias con un total de 417 posiciones. Varias secuencias utilizadas corresponden a la colección de referencia de Benjamin Victor, elaborada a partir de adultos morfológicamente caracterizados. El árbol filogenético, con la ubicación de los ejemplares larvarios dentro de cada clado específico, se presenta en la Figura 10.
Figura 8. Ejemplar de Labrisomus kalisherae, 176L, 4,30mm de LT, identificado por código de barras de ADN.
Figura 9. Ejemplares de Malacoctenus triangulatus identificados por código de barras de ADN. A = 374L, 2,84mm LT. B = 459L, 4,40mm LT. C = 474L, 4,55mm LT. D = 621L, 3,50mm LT. E = 762, 3,5mm LT.
Figura 10. Árbol filogenético de la familia Labrisomidae construido con secuencias del gen COI. Los ejemplares provenientes de Las Caracas se destacan con un marcador coloreado, e igualmente se resalta a color el clado que los incluye.
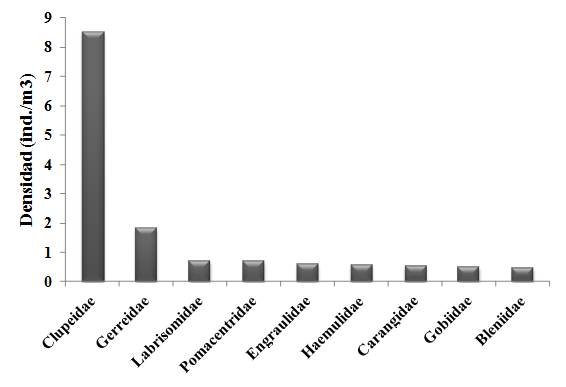
Estos resultados son relevantes ya que en la revisión realizada por Richards (2006) para los estados tempranos de vida en peces del Atlántico Occidental, el autor indica que a pesar de ser esta la familia con mayor número de especies en la zona, con 6 géneros y unas 49 especies, sólo había sido descrita para el momento la larva de Paraclinus marmoratus. Igualmente, en la mayoría de los trabajos realizados en el oriente venezolano sobre larvas de peces no aparece registrado este taxón. En nuestra investigación los labrisómidos se ubican como la tercera familia más abundante (Fig. 11), por lo que es probable que en trabajos previos estos peces hayan quedado dentro del grupo de larvas no identificadas o hayan sido objeto de determinaciones taxonómicas erróneas. Obviamente, los errores previos de identificación de larvas pertenecientes a esta familia son comprensibles por la ausencia de información al respecto.
Figura 11. Familias con mayor importancia numérica en las seis estaciones muestreadas y durante todo el periodo de estudio.
CONCLUSIONES
El código de barras del ADN resultó una herramienta importante en la identificación de estadios tempranos de peces.
La presencia de secuencias de adultos es crucial para lograr identificaciones de larvas y huevos.
Debido a la alta abundancia de larvas pertenecientes a Labrisomidae en este trabajo, y la frecuencia de adultos en la zona, es probable que exista un subregistro de estas etapas en investigaciones previas.
REFERENCIAS
Avise J. C. 2004. Molecular markers, natural history, and evolution. 2nd edition. Sinauer Associates, Sunderland, Massachusetts, USA.
Boltowskoy D. 1999. Zooplankton. South Atlantic. Backhuys Publishers, Leiden. The Netherlands. Vol. I y II. Mar de Plata, Argentina.
Felsenstein J. 1985. Confidence limits on phylogenies: An approach using the bootstrap. Evolution, 39:783-791.
Hall T. 2011. BioEdit: An important software for molecular biology. GERF Bulletin of Biosciences, 2(1):60-61.
Hebert P.D.N., A. Cywinska, S.L. Ball y J.R. deWaard. 2003. Biological identifications through DNA barcodes. Proc. R. Soc. Lond. B, 270, 313–321.
Holden M. y D. Raitt. 1975. Manual de ciencias pesqueras. Parte 2: Métodos para investigar los recursos y su aplicación. FAO, Nº 15.
Kimura M. 1980. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences. Journal of Molecular Evolution, 16:111-120.
Nelson, J.S., 1994. Fishes of the world. 3ª edición. John Wiley & Sons, Inc., New York. 600 p.
Novotny, V.; Basset, Y.; Miller, S.; Weiblen, G.; Bremer, B.; Cizek, L. & Drozd P. 2002. «Low host specificity of herbivorous insects in a tropical forest». Nature 416: pp. 841–844. doi: 10.1038/416841a (http://dx.doi.org/10.1038/416841a) Ratnasingham S. y P. D. N. Hebert. 2007. BOLD: The Barcode of Life Data System (www.barcodinglife.org). Molecular Ecology Notes, 7: 355–364.
Richards W.J. (Ed., 2006) Early stages of Atlantic fishes: an identification guide for western central North Atlantic. CRC Press, Boca Ratón, Florida, USA.
Saitou N. y M. Nei .1987. The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol., 4:406-425.
Tamura K., D. Peterson, N. Peterson, G. Stecher, M. Nei y S. Kumar. 2011. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molecular Biology and Evolution (submitted). doi:10.1093/molbev/msr121. <www.megasoftware.net>.
TODAS LAS IMÁGENES PUBLICADAS SON DE MI AUTORÍA Y PROPIEDAD. RESERVADOS TODOS LOS DERECHOS COMERCIALES.





¡Felicitaciones!
Estás participando para optar a la mención especial que se efectuará el domingo 14 de julio del 2019 a las 8:00 pm (hora de Venezuela), gracias a la cual el autor del artículo seleccionado recibirá la cantidad de 1 STEEM transferida a su cuenta.
¡También has recibido 1 ENTROKEN! El token del PROYECTO ENTROPÍA impulsado por la plataforma Steem-Engine.
Te participamos que puedes invertir en el PROYECTO ENTROPÍA mediante tu delegación de Steem Power y así comenzar a recibir ganancias de forma semanal transferidas automáticamente a tu monedero todos los lunes. Entra aquí para más información sobre cómo invertir en ENTROPÍA.
Contáctanos en Discord.
Apoya al trail de @Entropia y así podrás ganar recompensas de curación de forma automática. Entra aquí para más información sobre nuestro trail.
Puedes consultar el reporte diario de curación visitando @entropia.
Atentamente
El equipo de curación del PROYECTO ENTROPÍA
Muchas gracias por su apoyo.
Posted using Partiko iOS
This post has been voted on by the SteemSTEM curation team and voting trail. It is elligible for support from @curie and @utopian-io.
If you appreciate the work we are doing, then consider supporting our witness stem.witness. Additional witness support to the curie witness and utopian-io witness would be appreciated as well.
For additional information please join us on the SteemSTEM discord and to get to know the rest of the community!
Thanks for having added @steemstem as a beneficiary to your post. This granted you a stronger support from SteemSTEM.
Please consider using the steemstem.io app to get a stronger support.
Thanks for the support!!
Posted using Partiko iOS
Hi @afarina46!
Your post was upvoted by Utopian.io in cooperation with @steemstem - supporting knowledge, innovation and technological advancement on the Steem Blockchain.
Contribute to Open Source with utopian.io
Learn how to contribute on our website and join the new open source economy.
Want to chat? Join the Utopian Community on Discord https://discord.gg/h52nFrV
Thank you very much for your support, they have been very kind !!
Mil gracias por apoyarme!
Posted using Partiko iOS
Extraordinaria su publicación @afarina46, un trabajo que se deja leer con facilidad a pesar de no manejar el lenguaje técnico de ustedes los especialistas. Quedo complacida por este trabajo impecable. Gracias por la información. Va mi humilde apoyo, saludos.
Agradezco muchísimo sus comentarios. Su aporte es muy importante para mi.
Posted using Partiko iOS
Congratulations @afarina46! You have completed the following achievement on the Steem blockchain and have been rewarded with new badge(s) :
You can view your badges on your Steem Board and compare to others on the Steem Ranking
If you no longer want to receive notifications, reply to this comment with the word
STOPTo support your work, I also upvoted your post!
Thanks for the notification